这操作有点东西 (1)
来自好基友 Chaoli 的供稿支持!!!!
实际需求
有些同学做基因序列比对的时候,有如下图这样展示基因或蛋白序列差异位点的需求。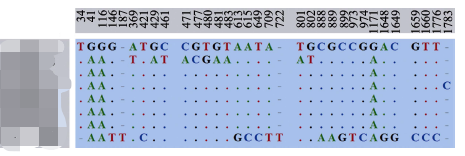
这时候我们一般是将多条基因放入一个 fasta 文件中,然后通过比对软件生成 multiple sequence alignment (MSA) 文件,再筛选出碱基差异位点的序列及碱基差异位点在 MSA 序列中原始位置的索引。
肉眼筛选这些位点比较麻烦并且容易出错,这里给大家提供一个简单的脚本自动提取差异位点及其索引。
MSA 文件一般可以使用MEGA软件比对后导出,或者使用 muscle 在线比对工具导出比对后的fasta格式文件。
脚本下载
https://www.dropbox.com/s/scm8pt4mfsypbtb/extract_diff_sites.exe?dl=0
点击链接,直接下载文件到本地,直接双击打开就可以使用了(仅支持 windows 平台)。
脚本使用
使用方法也非常简单,双击打开脚本后,按照提示选择比对好的 MSA 文件。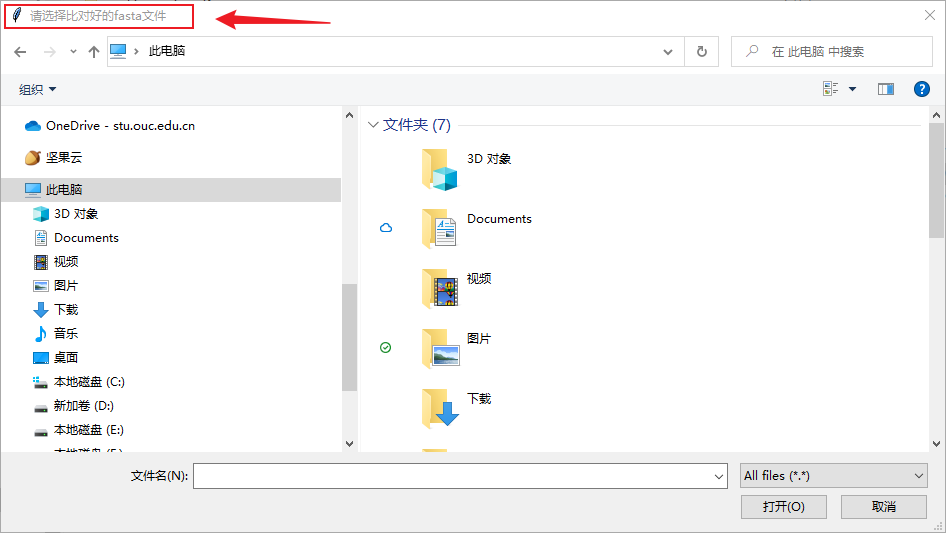
点击打开,脚本会自动提取碱基差异位点序列及其索引,弹出如下页面提示操作完成。
脚本所在目录会生成两个新文件。
一个是 Different_sites.fas fasta 格式文件。包含所有的碱基差异位点。
第二个是 sites_index.txt 文件,包含 Different_sites.fas 文件中碱基位点在原 MSA 文件中的位置索引信息。
这样 MSA 基因比对中的碱基差异位点提取就完成了。
RULE2: Treat yourself like someone you are responsible for helping. ——Jordan B. Peterson
评论
WalineTwikoo

